
Struttura, funzioni, proprietà, sintesi della citosina
Il citosina È una base azotata di tipo pirimidinico, utilizzata per la biosintesi di citidina-5'-monofosfato e deossicitidina-5'-monofosfato. Questi composti servono per la biosintesi, rispettivamente, dell'acido desossiribonucleico (DNA) e dell'acido ribonucleico (RNA). Il DNA immagazzina le informazioni genetiche e l'RNA ha varie funzioni.
Negli esseri viventi, la citosina non si trova in forma libera, ma forma comunemente ribonucleotidi o desossiribonucleotidi. Entrambi i tipi di composti hanno un gruppo fosfato, un ribosio e una base di azoto..
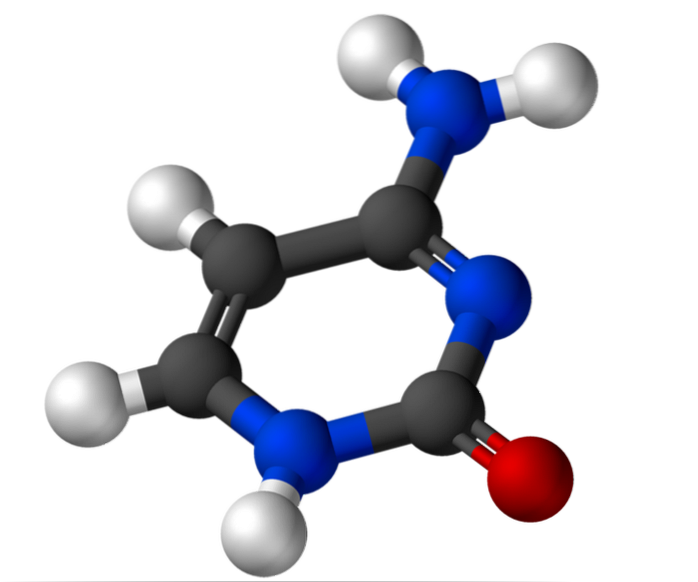
Il 2-carbonio del ribosio ha un gruppo idrossile (-OH) sui ribonucleotidi e un atomo di idrogeno (-H) sui desossiribonucleotidi. A seconda del numero di gruppi fosfato presenti, c'è citidina-5'-monofosfato (CMP), citidina-5'-difosfato (CDP) e citidina-5'-trifosfato (CTP).
Gli equivalenti deossigenati sono chiamati deossicitidina-5'-monofosfato (dCMP), deossicitidina-5'-difosfato (dCDP) e deossicitidina-5'-trifosfato (dCTP)..
La citosina, nelle sue varie forme, partecipa a diverse funzioni, come la biosintesi del DNA e dell'RNA, la biosintesi della glicoproteina e la regolazione dell'espressione genica..
Indice articolo
- 1 Struttura e proprietà
- 2 Biosintesi
- 2.1 Regolazione della biosintesi
- 2.2 Le pirimidine, come la citosina, vengono riciclate
- 3 Ruolo nella biosintesi del DNA
- 4 Ruolo nella stabilizzazione della struttura del DNA
- 5 Ruolo delle regioni ricche di citosina nel DNA
- 6 Ruolo nella biosintesi dell'RNA
- 7 Ruolo nella biosintesi della glicoproteina
- 8 Trattamenti chemioterapici contro la citosina e il cancro
- 9 Riferimenti
Struttura e proprietà
La citosina, 4-ammino-2-idrossipirimidina, ha la formula empirica C4H5N3O, il cui peso molecolare è di 111,10 g / mol, e viene purificato come polvere bianca.
La struttura della citosina è un anello eterociclico aromatico planare. La lunghezza d'onda della massima assorbanza (ʎmax) è a 260 nm. La temperatura di fusione della citosina supera i 300 ° C.
Per formare un nucleotide, la citosina è attaccata covalentemente, attraverso l'azoto 1, tramite un legame N-beta-glicosidico al carbonio 1 'del ribosio. Il carbonio 5 'viene esterificato con un gruppo fosfato.
Biosintesi
La biosintesi dei nucleotidi pirimidinici ha un percorso comune, costituito da sei fasi catalizzate da enzimi. Il percorso inizia con la biosintesi del carbamoil fosfato. Nei procarioti c'è un solo enzima: carbamoil fosfato sintasi. Questo è responsabile della sintesi di pirimidine e glutammina. Negli eucarioti sono presenti carbamoil fosfato sintasi I e II, responsabili, rispettivamente, della biosintesi della glutammina e delle pirimidine.
La seconda fase consiste nella formazione di N-carbamoilaspartato, da carboil fosfato e aspartato, una reazione catalizzata dall'aspartato transcabamoilasi (ATCasi).
Il terzo passaggio è la sintesi dell'L-diidrorotato, che provoca la chiusura dell'anello pirimidinico. Questo passaggio è catalizzato dalla diidrootasi..
La quarta fase è la formazione dell'orotato, che è una reazione redox catalizzata dalla diidroorotato deidrogenasi..
Il quinto passaggio consiste nella formazione di orotidilato (OMP) utilizzando fosforibosil pirofosfato (PRPP) come substrato e orotato fosforibosil transferasi come catalizzatore..
Il sesto passaggio è la formazione di uridilato (uridina-5'-monofosfato, UMP), una reazione catalizzata da una OMP-decarbossilasi..
Le fasi successive consistono nella fosforilazione di UMP, catalizzata dalle chinasi, per formare UTP, e il trasferimento di un gruppo amminico dalla glutammina a UTP per formare CTP, una reazione catalizzata dalla CTP sintetasi..
Regolazione della biosintesi
Nei mammiferi, la regolazione avviene a livello della carbamoil fosfato sintasi II, un enzima presente nel citosol, mentre la carbamoil fosfato sintasi I è mitocondriale.
La carbamoil fosfato sintasi II è regolata da un feedback negativo. I suoi regolatori, UTP e PRPP, sono, rispettivamente, inibitori e attivatori di questo enzima.
Nei tessuti non epatici, la carbamoil fosfato sintasi II è l'unica fonte di carbamoil fosfato. Mentre nel fegato, in condizioni di eccesso di ammoniaca, la carbamoil fosfato sintasi I produce, nei mitocondri, carbamoil fosfato, che viene trasportato al citosol, da dove entra nella via di biosintesi della pirimidina..
Un altro punto di regolazione è la OMP-decarbossilasi, che è regolata dall'inibizione competitiva. Il suo prodotto di reazione, UMP, compete con OMP per il sito di legame sulla OMP-decarbossilasi..
Le pirimidine, come la citosina, vengono riciclate
Il riciclo delle pirimidine ha la funzione di riutilizzare le pirimidine senza necessità di biosintesi de novo ed evitando la via degradativa. La reazione di riciclaggio è catalizzata dalla pirimimidina fosforibosiltransferasi. La reazione generale è la seguente:
Pirimidina + PRPP -> pirimidina nucleoside 5'-monofosfato + PPi
Nei vertebrati, la pirimimidina fosforibosiltransferasi si trova negli eritrociti. Le pirimidine substrato per questo enzima sono uracile, timina e orotato. La citosina viene riciclata indirettamente dall'uridina-5'-monofosfato.
Ruolo nella biosintesi del DNA
Durante la replicazione del DNA, le informazioni contenute nel DNA vengono copiate nel DNA da una DNA polimerasi..
La biosintesi dell'RNA richiede deossinucleotide trifosfato (dNTP), vale a dire: deossitimidina trifosfato (dTTP), deossicitidina trifosfato (dCTP), deossiadenina trifosfato (dATP) e deossiguanina trifosfato (dGTP). La reazione è:
(DNA)n residui + dNTP -> (DNA)n + 1 residuo + PPi
L'idrolisi del pirofosfato inorganico (PPi) fornisce l'energia per la biosintesi dell'RNA.
Ruolo nella stabilizzazione della struttura del DNA
Nella doppia elica del DNA, una purina a un filamento è collegata alla pirimidina a filamento opposto da legami idrogeno. Pertanto, la citosina è sempre collegata alla guanina da tre legami idrogeno: l'adenina è collegata alla timina da due legami idrogeno..
I legami idrogeno si rompono quando una soluzione di DNA nativo purificato, a pH 7, viene sottoposta a temperature superiori a 80 ºC. Ciò fa sì che la doppia elica del DNA formi due filamenti separati. Questo processo è noto come denaturazione..
La temperatura alla quale il 50% del DNA viene denaturato è nota come temperatura di fusione (Tm). Le molecole di DNA il cui rapporto tra guanina e citosina è superiore a quello di timina e adenina hanno valori di Tm più elevati rispetto a quelle il cui rapporto di basi è inverso.
Quanto sopra descritto costituisce la prova sperimentale che un maggior numero di legami idrogeno stabilizzano meglio le molecole di DNA native..
Ruolo delle regioni ricche di citosina nel DNA
Recentemente, è stato scoperto che il DNA del nucleo delle cellule umane può adottare strutture di motivi intervallati (iM). Queste strutture si verificano nelle regioni ricche di citosina.
La struttura iM consiste di quattro filamenti di DNA, a differenza del classico DNA a doppia elica che ha due filamenti. Più specificamente, due catene duplex parallele sono intervallate in un orientamento antiparallelo e sono tenute insieme da una coppia di citosine emiprotonate (C: C+).
Nel genoma umano, le strutture iM si trovano in regioni come promotori e telomeri. Il numero di strutture iM è maggiore durante la fase G1 / S del ciclo cellulare, in cui la trascrizione è elevata. Queste regioni sono siti di riconoscimento delle proteine coinvolti nell'attivazione del meccanismo trascrizionale..
D'altra parte, nelle regioni ricche di coppie di basi di guanina consecutive (C), il DNA tende ad assumere la forma dell'elica A, in condizioni di disidratazione. Questa forma è tipica dell'RNA e delle doppie bande DNA-RNA miste durante la trascrizione e la replicazione e in determinati momenti in cui il DNA è legato alle proteine..
È stato dimostrato che regioni di base consecutive della citosina creano un cerotto elettropositivo nella fessura principale del DNA. Pertanto, si ritiene che queste regioni si leghino alle proteine, predisponendo alcune regioni genomiche alla fragilità genetica..
Ruolo nella biosintesi dell'RNA
Durante la trascrizione, le informazioni contenute nel DNA vengono copiate nell'RNA da una RNA polimerasi. La biosintesi dell'RNA richiede nucleoside trifosfato (NTP), vale a dire: citidina trifosfato (CTP), uridina trifosfato (UTP), adenina trifosfato (ATP) e guanina trifosfato (GTP). La reazione è:
(RNA)n residui + NTP -> (RNA)n + 1 residuo + PPi
L'idrolisi del pirofosfato inorganico (PPi) fornisce l'energia per la biosintesi dell'RNA.
Ruolo nella biosintesi della glicoproteina
Il trasferimento sequenziale di esosi per formare oligosaccaridi, O-legati alle proteine, avviene da precursori nucleotidici.
Nei vertebrati, l'ultimo passaggio nella biosintesi degli oligosaccaridi legati all'O consiste nell'aggiunta di due residui di acido sialico (N-acetilneuraminico) da un precursore della citidina-5'-monofosfato (CMP). Questa reazione si verifica nel sacco trans Golgi.
Citosina e trattamenti chemioterapici antitumorali
L'acido tetraidrofolato (FH4) è una fonte di gruppi -CH3, ed è necessario per la biosintesi di dTMP da dUMP. Inoltre, si forma FH2. La riduzione di FH2 in FH4 richiede una riduttasi di folato e NADPH. Alcuni inibitori della folato reduttasi, come l'aminopterina e il metotrexato, vengono utilizzati nei trattamenti contro il cancro.
Il metotrexan è un inibitore competitivo. La folato reduttasi si lega con un'affinità 100 volte maggiore a questo inibitore che al suo substrato. L'aminopterina funziona in modo simile.
L'inibizione della folato reduttasi ostacola indirettamente la biosintesi della dTMP, e quindi quella della dCTP. L'inibizione diretta si verifica tramite gli inibitori dell'enzima timidilato sintetasi, che catalizza dTMP da dUMP. Questi inibitori sono il 5-fluorouracile e la 5-fluoro-2-deossiuridina..
Ad esempio, il 5-fluoroacile non è di per sé un inibitore ma viene prima convertito, nel percorso di riciclaggio, in desossiuridina mphosphate d (FdUMP), che si lega e inibisce la timidilato sintetasi..
Sostanze analoghe alla glutammina, azaserina e acivicina, inibiscono la glutammina amidotransferasi. L'azarin è stata una delle prime sostanze scoperte che agiscono come inattivatore del suicidio.
Riferimenti
- Assi, H.A., Garavís, M., González, C. e Damha, M.J. 2018. i-Motif DNA: caratteristiche strutturali e significato per la biologia cellulare. Nuclei Acids Research, 46: 8038-8056.
- Bohinski, R. 1991. Biochemistry. Addison-Wesley Iberoamericana, Wilmington, Delaware.
- Devlin, T.M. 2000. Biochimica. Editoriale Reverté, Barcellona.
- Lodish, H., Berk, A., Zipurski, S.L., Matsudaria, P., Baltimore, D., Darnell, J. 2003. Biologia cellulare e molecolare. Editoriale Medica Panamericana, Buenos Aires, Bogotá, Caracas, Madrid, Messico, Sāo Paulo.
- Nelson, D. L., Cox, M. M. 2008. Lehninger-Principles of biochemistry. W.H. Freeman, New York.
- Voet, D. e Voet, J. 2004. Biochemistry. John Wiley and Sons, Stati Uniti.


Nessun utente ha ancora commentato questo articolo.