
Traduzione del processo del DNA in eucarioti e procarioti

Il Traduzione del DNA è il processo mediante il quale l'informazione contenuta negli RNA messaggeri prodotti durante la trascrizione (la copia dell'informazione in una sequenza di DNA sotto forma di RNA) viene "tradotta" in una sequenza amminoacidica mediante sintesi proteica.
Da una prospettiva cellulare, l'espressione genica è una faccenda relativamente complessa che avviene in due fasi: trascrizione e traduzione..
Tutti i geni che sono espressi (indipendentemente dal fatto che codificano o meno sequenze peptidiche, cioè proteine) lo fanno inizialmente trasferendo le informazioni contenute nella loro sequenza di DNA a una molecola di RNA messaggero (mRNA) attraverso un processo chiamato trascrizione.
La trascrizione è ottenuta da speciali enzimi noti come RNA polimerasi, che utilizzano uno dei filamenti complementari del DNA del gene come modello per la sintesi di una molecola "pre-mRNA", che viene successivamente elaborata per formare un mRNA maturo..
Per i geni che codificano per proteine, le informazioni contenute negli mRNA maturi vengono “lette” e tradotte in amminoacidi secondo il codice genetico, che specifica quale codone o tripletto nucleotidico corrisponde a quale particolare amminoacido..
La specificazione della sequenza amminoacidica di una proteina, quindi, dipende dalla sequenza iniziale di basi azotate nel DNA che corrisponde al gene e quindi nell'mRNA che trasporta questa informazione dal nucleo al citosol (nelle cellule eucariotiche); processo che è anche definito come sintesi proteica guidata da mRNA.
Dato che ci sono 64 possibili combinazioni delle 4 basi azotate che compongono DNA e RNA e solo 20 aminoacidi, un amminoacido può essere codificato da diverse triplette (codoni), motivo per cui il codice genetico si dice "degenere" (ad eccezione dell'amminoacido metionina, che è codificato da un codone AUG unico).
Indice articolo
- 1 Traduzione eucariotica (processo a fasi)
- 1.1 - Elaborazione degli mRNA prima della loro traduzione
- 1.2 - Ribosomi
- 1.3 - Inizia la traduzione
- 2 Traduzione procariotica (fasi-processi)
- 2.1 Processo
- 3 Riferimenti
Traduzione eucariotica (processo a fasi)
Nelle cellule eucariotiche la trascrizione avviene nel nucleo e la traduzione nel citosol, quindi anche gli mRNA che si formano durante il primo processo giocano un ruolo nel trasporto delle informazioni dal nucleo al citosol, dove si trovano le cellule. (ribosomi).
È importante ricordare che la compartimentazione della trascrizione e della traduzione negli eucarioti è vera per il nucleo, ma non è la stessa per gli organelli con il proprio genoma come i cloroplasti e i mitocondri, che hanno sistemi più simili a quelli degli organismi procarioti..
Le cellule eucariotiche hanno anche ribosomi citosolici attaccati alle membrane del reticolo endoplasmatico (reticolo endoplasmatico ruvido), in cui avviene la traduzione di proteine che sono destinate ad inserirsi nelle membrane cellulari o che richiedono elaborazioni post-traduzionali che avvengono in detto compartimento..
- Elaborazione degli mRNA prima della loro traduzione
Gli mRNA vengono modificati alle loro estremità man mano che vengono trascritti:
- Quando l'estremità 5 'dell'mRNA emerge dalla superficie della RNA polimerasi II durante la trascrizione, viene immediatamente "attaccata" da un gruppo di enzimi che sintetizzano una "cappa" composta da 7-metil guanilato e che è collegata al nucleotide terminale . dell'mRNA tramite un legame trifosfato 5 ', 5'.
- L'estremità 3 'dell'mRNA subisce una "scissione" da parte di un'endonucleasi, che genera un gruppo idrossile libero 3' a cui è attaccata una "stringa" o "coda" di residui di adenina (da 100 a 250), a cui viene aggiunto uno allo stesso tempo da un enzima poli (A) polimerasi.
Il "cappuccio 5 '" e la "coda poli A ”svolgono funzioni nella protezione delle molecole di mRNA contro la degradazione e, inoltre, funzionano rispettivamente nel trasporto di trascritti maturi verso il citosol e nell'inizio e fine della traduzione, rispettivamente..
Corte e giuntare
Dopo la trascrizione, gli mRNA "primari" con le loro due estremità modificate, ancora presenti nel nucleo, subiscono un processo di "splicing" mediante il quale le sequenze introniche vengono generalmente rimosse e gli esoni risultanti vengono uniti (elaborazione post-trascrizionale), con cui vengono ottenuti che lasciano il nucleo e raggiungono il citosol.
La giunzione viene eseguita da un complesso riboproteico chiamato spliceosome (Anglicismo di spliceosome), costituito da cinque piccole ribonucleoproteine e molecole di RNA, in grado di “riconoscere” le regioni da rimuovere dal trascritto primario.
In molti eucarioti esiste un fenomeno noto come "splicing alternativo", il che significa che diversi tipi di modifiche post-trascrizionali possono produrre diverse proteine o isoenzimi che differiscono tra loro in alcuni aspetti delle loro sequenze..
- Ribosomi
Quando i trascritti maturi lasciano il nucleo e vengono trasportati per la traduzione nel citosol, vengono elaborati dal complesso traslazionale noto come ribosoma, che consiste in un complesso di proteine associate alle molecole di RNA..
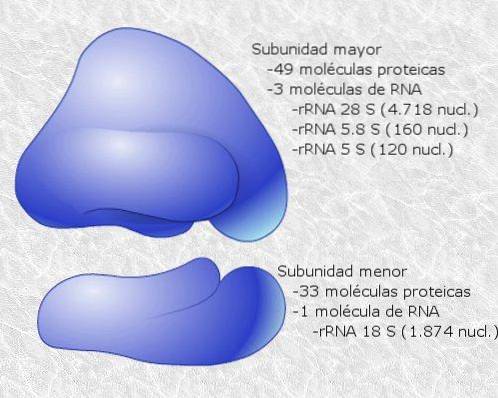
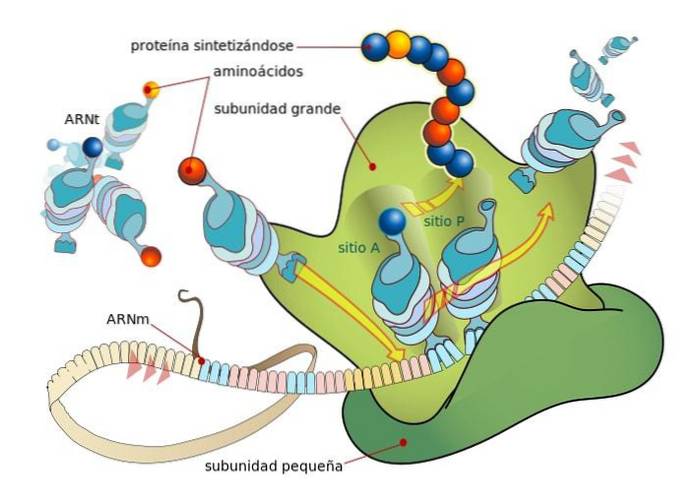
I ribosomi sono composti da due subunità, una "grande" e l'altra "piccola", che si dissociano liberamente nel citosol e si legano o si associano alla molecola di mRNA che viene tradotta.
Il legame tra ribosomi e mRNA dipende da molecole di RNA specializzate che si associano a proteine ribosomiali (RNA ribosomiale o rRNA e RNA di trasferimento o tRNA), ognuna delle quali ha funzioni specifiche..
I TRNA sono "adattatori" molecolari, poiché attraverso una delle loro estremità possono "leggere" ogni codone o tripletta nell'mRNA maturo (per complementarità di basi) e attraverso l'altra possono legarsi all'amminoacido codificato dal codone "letto"..
Le molecole di rRNA, d'altra parte, sono responsabili dell'accelerazione (catalizzazione) del processo di legame di ciascun amminoacido nella catena peptidica nascente.
Un mRNA eucariotico maturo può essere "letto" da molti ribosomi, tante volte quante sono indicate dalla cellula. In altre parole, lo stesso mRNA può dare origine a molte copie della stessa proteina..
Codone iniziale e frame di lettura
Quando un mRNA maturo viene avvicinato da subunità ribosomiali, il complesso riboproteico "scansiona" la sequenza di detta molecola fino a trovare un codone di inizio, che è sempre AUG e comporta l'introduzione di un residuo di metionina.
Il codone AUG definisce il frame di lettura per ogni gene e, inoltre, definisce il primo amminoacido di tutte le proteine tradotte in natura (questo amminoacido viene spesso eliminato post-traduzionale).
Fermare i codoni
Altri tre codoni sono stati identificati come quelli che inducono la terminazione della traduzione: UAA, UAG e UGA..
Quelle mutazioni che comportano un cambiamento delle basi azotate nella tripletta che codifica per un amminoacido e che danno luogo a codoni di stop sono note come mutazioni senza senso, poiché provocano un arresto prematuro del processo di sintesi, che forma proteine più corte.
Regioni non tradotte
Vicino all'estremità 5 'delle molecole di mRNA maturo ci sono regioni che non sono tradotte (UTR). Regione non tradotta), chiamate anche sequenze "leader", che si trovano tra il primo nucleotide e il codone di inizio della traduzione (AUG).
Queste regioni UTR che non sono tradotte hanno siti specifici per il legame con i ribosomi e nell'uomo, ad esempio, sono lunghi circa 170 nucleotidi, tra i quali ci sono regioni regolatrici, siti di legame proteico che funzionano nella regolazione della traduzione, ecc..
- Inizio della traduzione
La traduzione, così come la trascrizione, si compone di 3 fasi: una fase di inizio, una fase di allungamento e infine una fase di terminazione..
Iniziazione
Consiste nell'assemblaggio del complesso traslazionale sull'mRNA, che merita l'unione di tre proteine note come fattori di iniziazione (IF). Fattore di iniziazione) IF1, IF2 e IF3 alla piccola subunità del ribosoma.
Il complesso "pre-iniziazione" formato dai fattori di iniziazione e la piccola subunità ribosomiale, a sua volta, si lega con un tRNA che "trasporta" un residuo di metionina e questo insieme di molecole si lega all'mRNA, vicino al codone di inizio. AUG.
Questi eventi portano al legame dell'mRNA alla grande subunità ribosomiale, portando al rilascio di fattori di iniziazione. La grande subunità ribosomiale ha 3 siti di legame per le molecole di tRNA: il sito A (amminoacido), il sito P (polipeptide) e il sito E (uscita)..
Il sito A si lega all'anticodone dell'amminoacil-tRNA che è complementare a quello dell'mRNA che viene tradotto; il sito P è dove l'amminoacido viene trasferito dal tRNA al peptide nascente e il sito E è dove si trova nel tRNA "vuoto" prima di essere rilasciato nel citosol dopo che l'amminoacido è stato erogato.
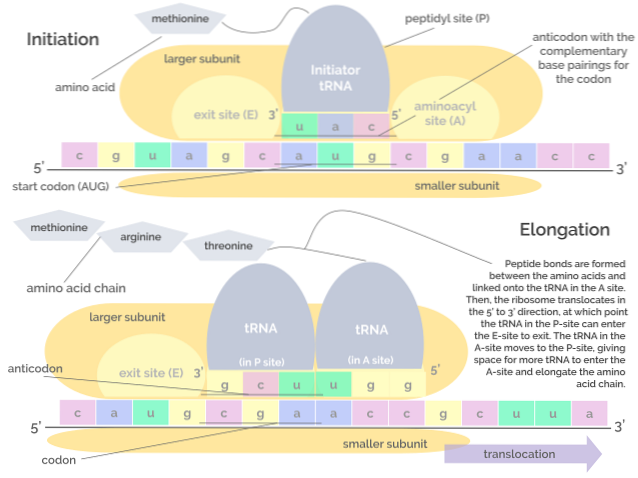
Allungamento
Questa fase consiste nel “movimento” del ribosoma lungo la molecola di mRNA e nella traduzione di ogni codone che si “legge”, il che implica la crescita o l'allungamento della catena polipeptidica alla nascita..
Questo processo richiede un fattore noto come fattore di allungamento G ed energia sotto forma di GTP, che è ciò che guida la traslocazione dei fattori di allungamento lungo la molecola di mRNA mentre viene tradotta..
L'attività della peptidil transferasi degli RNA ribosomiali consente la formazione di legami peptidici tra amminoacidi successivi che vengono aggiunti alla catena.
Risoluzione
La traduzione termina quando il ribosoma incontra uno dei codoni di terminazione, poiché i tRNA non riconoscono questi codoni (non codificano gli amminoacidi). Si legano anche proteine note come fattori di rilascio, che facilitano il distacco dell'mRNA dal ribosoma e la dissociazione delle sue subunità..
Traduzione procariotica (passaggi-processi)
Nei procarioti, come nelle cellule eucariotiche, i ribosomi responsabili della sintesi proteica si trovano nel citosol (il che vale anche per il macchinario trascrizionale), fatto che consente il rapido aumento della concentrazione citosolica di una proteina quando l'espressione dei geni quella codifica aumenta.
Sebbene non sia un processo estremamente comune in questi organismi, gli mRNA primari prodotti durante la trascrizione possono subire una maturazione post-trascrizionale attraverso lo "splicing". Tuttavia, il più comune è osservare i ribosomi attaccati al trascritto primario che lo stanno traducendo nello stesso momento in cui viene trascritto dalla sequenza di DNA corrispondente..
In considerazione di quanto sopra, la traduzione in molti procarioti inizia all'estremità 5 ', poiché l'estremità 3' dell'mRNA rimane attaccata al DNA stampo (e si verifica in concomitanza con la trascrizione)..
Regioni non tradotte
Le cellule procariotiche producono anche mRNA con regioni non tradotte note come "scatola di Shine-Dalgarno" e la cui sequenza di consenso è AGGAGG. Come è evidente, le regioni UTR dei batteri sono notevolmente più corte di quelle delle cellule eucariotiche, sebbene esercitino funzioni simili durante la traduzione..
Processi
Nei batteri e in altri organismi procarioti il processo di traduzione è abbastanza simile a quello delle cellule eucariotiche. Consiste inoltre di tre fasi: inizio, allungamento e terminazione, che dipendono da fattori procariotici specifici, diversi da quelli utilizzati dagli eucarioti..
L'allungamento, ad esempio, dipende da fattori di allungamento noti come EF-Tu e EF-Ts, piuttosto che dal fattore G degli eucarioti..
Riferimenti
- Alberts, B., Johnson, A., Lewis, J., Raff, M., Roberts, K. e Walter, P. (2007). Biologia molecolare della cellula. Garland Science. New York, 1392.
- Clancy, S. & Brown, W. (2008) Traduzione: DNA to mRNA to Protein. Educazione alla natura 1 (1): 101.
- Griffiths, A. J., Wessler, S. R., Lewontin, R. C., Gelbart, W. M., Suzuki, D. T. e Miller, J. H. (2005). Un'introduzione all'analisi genetica. Macmillan.
- Lodish, H., Berk, A., Kaiser, C. A., Krieger, M., Scott, M. P., Bretscher, A.,… e Matsudaira, P. (2008). Biologia cellulare molecolare. Macmillan.
- Nelson, D. L., Lehninger, A. L., & Cox, M. M. (2008). Principi di biochimica di Lehninger. Macmillan.
- Rosenberg, L. E., & Rosenberg, D. D. (2012). Geni e genomi umani: scienza. Salute, società, 317-338.


Nessun utente ha ancora commentato questo articolo.