
Struttura quaternaria delle proteine caratteristiche
Il struttura quaternaria di proteine definisce le relazioni spaziali tra ciascuna delle sue subunità polipeptidiche unite da forze non covalenti. Nelle proteine polimeriche, ciascuna delle catene polipeptidiche che la compongono è chiamata subunità o protomeri.
Le proteine possono essere costituite da uno (monomerico), due (dimerico), diversi (oligomerici) o molti protomeri (polimerici). Questi protomeri possono avere una struttura molecolare simile o molto diversa. Nel primo caso si dice che siano proteine omotipiche e nel secondo caso, eterotipiche.
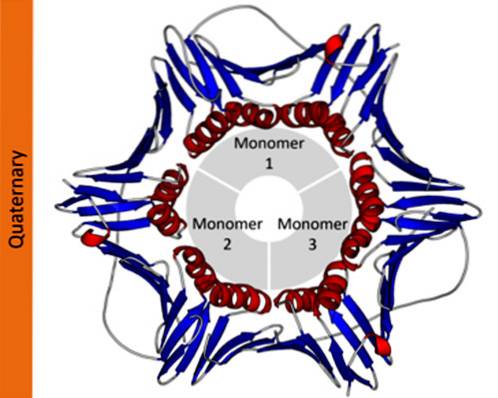
Nella notazione scientifica, i biochimici usano lettere greche in pedice per descrivere la composizione protomerica delle proteine. Ad esempio, una proteina omotipica tetramerica è designata come α4, mentre una proteina tetramerica composta da due diversi dimeri è designata come αDueβDue.
Indice articolo
- 1 Struttura proteica
- 1.1 Struttura primaria
- 1.2 Struttura secondaria
- 1.3 Struttura terziaria
- 1.4 Struttura quaternaria
- 2 Stabilità della struttura quaternaria
- 2.1 Interazioni idrofobiche
- 2.2 Interazioni di Van der Waals
- 2.3 Interazioni carico-carico
- 2.4 Legami idrogeno
- 2.5 Interazioni dei dipoli
- 3 Interazioni tra protomeri
- 3.1 Interazioni omotipiche
- 3.2 Interazioni eterotipiche
- 4 Riferimenti
Struttura delle proteine
Le proteine sono molecole complesse che assumono diverse configurazioni tridimensionali. Queste configurazioni sono uniche per ciascuna proteina e consentono loro di svolgere funzioni molto specifiche. I livelli di organizzazione strutturale delle proteine sono i seguenti.
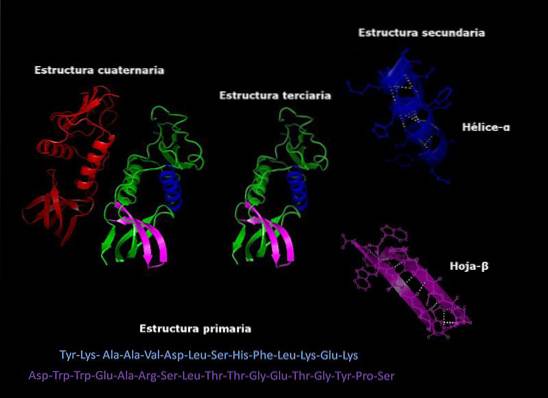
Struttura primaria
Si riferisce alla sequenza in cui i diversi amminoacidi sono disposti nella catena polipeptidica. Questa sequenza è data dalla sequenza di DNA che codifica detta proteina.
Struttura secondaria
La maggior parte delle proteine non sono lunghe catene completamente estese di amminoacidi, ma piuttosto hanno regioni che vengono piegate regolarmente in eliche o fogli. Questa piegatura è quella che riceve il nome di struttura secondaria.
Struttura terziaria
Le aree piegate della struttura secondaria possono, a loro volta, essere piegate e assemblate in strutture più compatte. Quest'ultima piega è ciò che conferisce alla proteina la sua forma tridimensionale..
Struttura quaternaria
Nelle proteine che sono costituite da più di una subunità, le strutture quaternarie sono le relazioni spaziali che esistono tra ciascuna subunità, che sono collegate da legami non covalenti..
Stabilità della struttura quaternaria
La struttura tridimensionale delle proteine è stabilizzata da interazioni deboli o non covalenti. Sebbene questi legami o interazioni siano molto più deboli dei normali legami covalenti, sono numerosi e il loro effetto cumulativo è potente. Qui vedremo alcune delle interazioni più comuni.
Interazioni idrofobiche
Alcuni amminoacidi contengono catene laterali idrofobiche. Quando le proteine hanno questi amminoacidi, il ripiegamento della molecola ordina queste catene laterali verso l'interno della proteina e le protegge dall'acqua. La natura delle diverse catene laterali significa che contribuiscono in modi diversi all'effetto idrofobo..
Interazioni di Van der Waals
Queste interazioni si verificano quando le molecole o gli atomi che non sono legati da legami covalenti si avvicinano troppo e per questo motivo i loro orbitali elettronici più esterni iniziano a sovrapporsi..
In quel momento si instaura una forza repulsiva tra questi atomi che cresce molto rapidamente man mano che i rispettivi centri si avvicinano. Queste sono le cosiddette "forze di van der Waals".
Interazioni carico-carico
È l'interazione elettrostatica che si verifica tra una coppia di particelle cariche. Nelle proteine si verifica questo tipo di interazione, sia per la carica elettrica netta della proteina, sia per la carica individuale degli ioni in essa contenuti. Questo tipo di interazione è talvolta chiamato ponte salato..
Legami di idrogeno
Viene stabilito un legame idrogeno tra un atomo di idrogeno legato in modo covalente a un gruppo donatore di legami idrogeno e una coppia di elettroni liberi appartenenti a un gruppo accettore di legame.
Questo tipo di legame è molto importante, poiché le proprietà di molte molecole, comprese quelle dell'acqua e delle molecole biologiche, sono in gran parte dovute ai legami idrogeno. Condivide le proprietà dei legami covalenti (gli elettroni sono condivisi) e anche delle interazioni non covalenti (interazione carica-carica).
Interazioni dipolari
Nelle molecole, comprese le proteine, che non presentano una carica netta, può verificarsi una disposizione non uniforme delle loro cariche interne, con un estremo leggermente più negativo dell'altro. Questo è ciò che è noto come dipolo.
Questa condizione dipolare della molecola può essere permanente, ma può anche essere indotta. I dipoli possono essere attratti da ioni o altri dipoli. Se i dipoli sono permanenti, l'interazione ha una portata maggiore di quella che si verifica con i dipoli indotti.
Oltre a queste interazioni non covalenti, alcune proteine oligomeriche stabilizzano la loro struttura quaternaria attraverso un tipo di legame covalente, il legame disolfuro. Questi sono stabiliti tra i gruppi sulfidrilici delle cisteine di diversi protomeri.
I legami disolfuro aiutano anche a stabilizzare la struttura secondaria delle proteine, ma in questo caso collegano i residui cisteinilici all'interno dello stesso polipeptide (legami disolfuro intrapolipeptidici)..
Interazioni tra protomeri
Come notato sopra, nelle proteine che sono costituite da più subunità o protomeri, queste subunità possono essere simili (omotipiche) o diverse (eterotipiche)..
Interazioni omotipiche
Le subunità che compongono una proteina sono catene polipeptidiche asimmetriche. Tuttavia, nelle interazioni omotipiche, queste subunità possono associarsi in modi diversi, ottenendo diversi tipi di simmetria..
I gruppi interagenti di ciascun protomero si trovano generalmente in posizioni diverse, motivo per cui sono chiamati interazioni eterologhe. Le interazioni eterologhe tra le diverse subunità, talvolta avvengono in modo tale che ciascuna subunità sia attorcigliata rispetto alla precedente, potendo realizzare una struttura elicoidale.
In altre occasioni le interazioni avvengono in modo tale che gruppi definiti di subunità sono disposti attorno a uno o più assi di simmetria, in quella che è nota come simmetria di gruppi di punti. Quando ci sono più assi di simmetria, ogni subunità ruota rispetto al suo vicino di 360 ° / n (dove n rappresenta il numero di assi).
Tra i tipi di simmetria che si ottengono in questo modo ci sono, ad esempio, elicoidale, cubica e icosaedrica.
Quando due subunità interagiscono attraverso un asse binario, ciascuna unità ruota di 180 ° rispetto all'altra, attorno a quell'asse. Questa simmetria è nota come simmetria C.Due. In esso, i siti di interazione in ciascuna subunità sono identici; in questo caso non si parla di interazione eterologa, ma di interazione isologa.
Se invece l'associazione tra le due componenti del dimero è eterologa, si otterrà un dimero asimmetrico.
Interazioni eterotipiche
Le subunità interagenti in una proteina non sono sempre della stessa natura. Ci sono proteine che sono composte da dodici o più subunità differenti.
Le interazioni che mantengono la stabilità della proteina sono le stesse delle interazioni omotipiche, ma generalmente si ottengono molecole completamente asimmetriche..
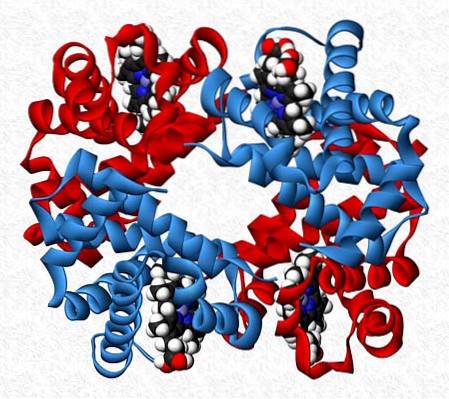
L'emoglobina, ad esempio, è un tetramero che ha due coppie di diverse subunità (αDueβDue).
Riferimenti
- C.K. Mathews, K.E. van Holde & K.G. Ahern (2002). Biochimica. 3a edizione. Benjamin / Cummings Publishing Company, Inc.
- R.K. Murray, P. Mayes, D.C. Granner & V.W. Rodwell (1996). Biochimica di Harper. Appleton e Lange
- J.M. Berg, J.L. Tymoczko e L. Stryer (2002). Biochimica. 5a edizione. W. H. Freeman and Company.
- J. Koolman e K.-H. Roehm (2005). Atlante dei colori di biochimica. 2a edizione. Thieme.
- A. Lehninger (1978). Biochimica. Ediciones Omega, S.A.
- L. Stryer (1995). Biochimica. W.H. Freeman and Company, New York.



Nessun utente ha ancora commentato questo articolo.