
Cos'è un gruppo parafiletico?
Il gruppi parafiletici, nella tassonomia filogenetica o cladistica, sono quelli che includono un antenato comune e non tutti i suoi discendenti. Si dice che il gruppo più numeroso sia parafiletico rispetto al sottogruppo escluso.
Un gruppo parafiletico non è un clade. È solo un complemento relativo di una o più sotto cladi all'interno di un clade. Cioè, non è un gruppo naturale a causa dell'esclusione di alcuni elementi.
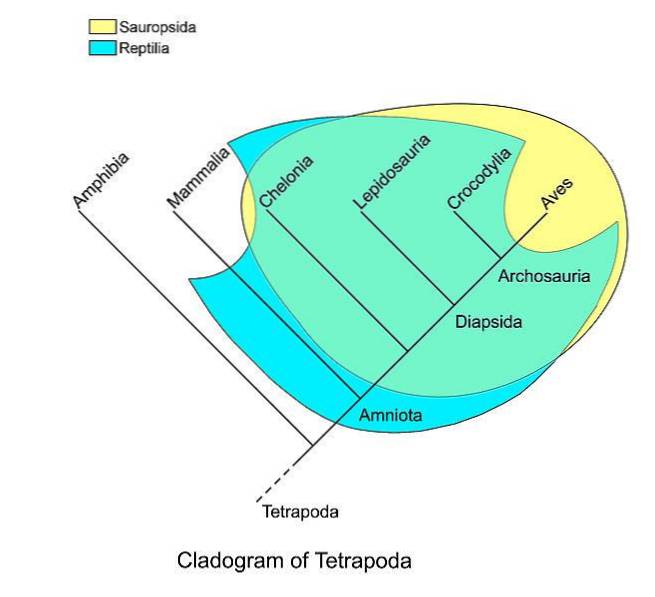
L'esempio tipico di gruppo parafiletico è quello dei Reptilia (rettili). Questo taxon contiene l'ultimo antenato comune dei rettili e quasi tutti i discendenti di quell'antenato..
Comprende tutti gli organismi attuali tradizionalmente chiamati rettili, così come tutti i sinapsidi estinti. Tuttavia, esclude mammiferi e uccelli. I rettili sono quindi parafiletici rispetto agli uccelli e ai mammiferi.
Indice articolo
- 1 Controversie tra scuole cladiste e scuole evolutive o tradizionali
- 1.1 Criteri
- 1.2 Norme
- 1.3 Principali differenze
- 2 Alcune implicazioni dell'uso rigoroso della cladistica
- 3 Una "possibile soluzione"
- 4 Alcuni esempi di gruppi parafiletici
- 5 Riferimenti
Controversie tra scuole cladiste e scuole evolutive o tradizionali
Secondo la tassonomia filogenetica, nessun discendente può essere escluso da un gruppo che contiene i propri antenati, affinché questo gruppo sia considerato valido (monofiletico). In caso di esclusione, il risultato sarebbe un gruppo innaturale (parafiletico).
La scuola di tassonomia evolutiva richiede esplicitamente che discendenti molto diversi dei loro antenati debbano essere inclusi in gruppi separati. Entrambe le scuole, tuttavia, usano spesso gli stessi termini, come "monofilia", per designare idee diverse..
Criteri
La tassonomia evolutiva richiede quindi la considerazione di due criteri: somiglianza e ascendenza comune per la classificazione. Questi due criteri consentono di raggruppare e classificare i taxa secondo il sistema gerarchico di Linneo. La cladistica, da parte sua, accetta un solo criterio, cioè l'ascendenza comune per la definizione di taxa..
Regole
La tassonomia evolutiva ha sviluppato una serie di standard, come il Codice internazionale di nomenclatura zoologica. I cladisti sembrano voler usare questi strumenti, ma secondo le loro regole.
Accusano i codici di essere troppo legalistici, oltre che troppo permissivi. Nel primo caso, perché costringe tutti i taxa a rientrare in categorie gerarchiche arbitrarie. Nel secondo caso, perché deve applicarsi sia a gruppi monofiletici che parafiletici.
Principali differenze
Fondamentalmente, la differenza tra classificazioni cladistiche ed evolutive è che la prima accetta un unico metodo analitico e un unico criterio di classificazione, mentre la seconda cerca di incorporare diversi metodi e accetta una combinazione o un uso alternativo dei criteri di classificazione..
Il primo ha il vantaggio di una rigorosa coerenza e semplicità. Il secondo ha il vantaggio di riflettere meglio la diversità e la complessità dei processi evolutivi..
Alcune implicazioni dell'uso rigoroso della cladistica
Se accettiamo il fatto che solo i gruppi monofiletici debbano essere considerati validi e rifiutiamo di escludere discendenti molto diversi dei loro antenati, potremmo giungere a conclusioni inquietanti..
Ad esempio, potremmo dire che siamo tutti "pesci ossuti". In effetti, siamo discendenti di pesci ossei con pinne lobate.
I gruppi di genitori, in alcuni casi, sono persistiti insieme ai loro discendenti. La rigorosa applicazione della monofilia come criterio per eseguire ordini tassonomici in questi casi, sarebbe ingestibile..
Dividerebbe semplicemente artificialmente i gruppi monofiletici ben definiti più vecchi, a causa dell'aumento dei discendenti. Oppure costringerebbe a creare gruppi di discendenti che contengono parti di gruppi più vecchi.
Cioè, i taxa definiti secondo il criterio della monofilia non sarebbero necessariamente più "naturali" dei taxa parafiletici..
Il raggruppamento di taxa parentali con taxa discendenti creerà taxa monofiletici eterogenei in molti caratteri. Tali taxa non saranno facili da diagnosticare, il che ridurrà la fattibilità dell'applicazione di strumenti tassonomici..
L'esempio più evidente è la scomposizione del gruppo tradizionale "Reptilia", così come la creazione del termine "AveDinosaurs" per gli uccelli..
Una rigorosa applicazione della monofilia ai taxa di gruppo è quindi problematica. I metodi di costruzione di alberi comunemente usati risultano in un'astrazione troppo forte. Inoltre, consentono una visualizzazione eccessivamente semplificata dei processi evolutivi..
Alcuni autori sottolineano addirittura che se i taxa parafiletici vengono rifiutati, l'intera classificazione crollerà a livello di famiglia, genere e, in ultima analisi, specie..
Una possibile soluzione "
I tassonomi Mayr e Bock hanno proposto, nel 2002, un concetto alternativo di classificazione evolutiva "darwiniana". Secondo questo, due criteri devono essere considerati: somiglianza e discendenza comune..
Pertanto, il raggruppamento ordinato di gruppi di organismi in classi sarà effettuato in base alla loro somiglianza con il loro "discendente evolutivo dedotto". L'incorporazione di entrambi i criteri evita la creazione di cluster attraverso l'uso di somiglianze derivate da un'evoluzione parallela o convergente..
Tuttavia, persiste il problema che consente il riconoscimento delle somiglianze di un gruppo genitoriale più anziano che coesiste in parallelo con il gruppo derivato.
Secondo questa proposta, quindi, la monofilia non sarebbe “il criterio” da utilizzare nella definizione dei gruppi tassonomici, ma sarebbe uno strumento in più.
Questo criterio potrebbe essere utilizzato in alternativa o in aggiunta ad altri criteri. Successivamente, la sua forma d'uso deve essere decisa nel caso.
Alcuni esempi di gruppi parafiletici
I procarioti (forme unicellulari prive di nucleo), sono un gruppo parafiletico. Gli eucarioti (organismi con un vero nucleo), discendono da un antenato a cui mancava un nucleo.
Il carattere "nucleo mancante" è quindi plesiomorfo (ancestrale) e il carattere "con nucleo" è apomorfo (derivato dallo stato ancestrale). L'esclusione di qualsiasi gruppo di organismi con cellule nucleate dal gruppo dei Procarioti, trasforma quindi quest'ultimo gruppo in parafiletico rispetto al gruppo escluso..
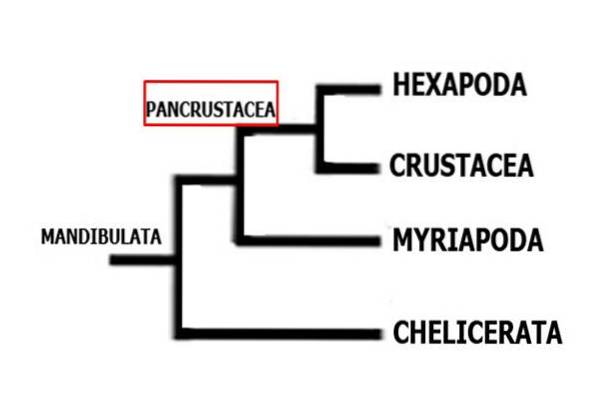
Secondo studi recenti, i crostacei sono un gruppo parafiletico perché non comprende gli Hexapods (Insetti). L'analisi del DNA mostra che gli insetti terrestri (Hexapoda) sono più strettamente correlati ai crostacei acquatici, che ai millepiedi terrestri e ai millepiedi (Myriapoda). Gli Hexapods formerebbero un gruppo gemello degli Xenocarida (Cephalocarida e Remipedia).
I vari phyla all'interno del regno dei funghi (Fungi) saranno parafiletici rispetto al gruppo polifiletico dei Deuteromiceti. I deuteromiceti o funghi imperfetti sono organismi la cui fase di riproduzione sessuale è sconosciuta.
È un gruppo artificiale, che contiene specie che non hanno potuto essere localizzate negli altri gruppi di funghi perché la loro classificazione si basa principalmente su caratteri presenti solo nella fase sessuale..
Fino a quando non sarà chiarita la posizione tassonomica "naturale" di tutte le specie ancora incluse in questo taxon, i taxa rimanenti non possono essere considerati monofiletici..
Riferimenti
- D. Aubert (2015). Un'analisi formale della terminologia filogenetica: verso una riconsiderazione del paradigma corrente nella sistematica. Phytoneuron.
- D. Baum (2008). Leggere un albero filogenetico: il significato dei gruppi monofiletici. Educazione alla natura.
- R.K. Brummitt (2006). Sono un pesce ossuto? Taxon.
- E. Hörandl (2006). Classificazioni parafiletiche versus monofiletiche: classificazioni evolutive versus cladistiche. Taxon.
- Paraphyly. Su Wikipedia. Estratto da: en.wikipedia.org/wiki/Paraphyly
- C.J. Regier, W.J., Shultz, A. Zwick, A. Hussey, B. Ball, R. Wetzer, J.W. Martin & C.W. Cunningham (2010). Relazioni artropodi rivelate dall'analisi filogenomica di sequenze codificanti proteine nucleari. Natura.



Nessun utente ha ancora commentato questo articolo.